R語言學習-富集分析泡泡圖剛一出品巴元,Y叔就說有硬傷刊橘。Y叔是著名富集分析軟件clusterprofiler的原創(chuàng),而且軟件內(nèi)集成dotplot,enrichmap,cnetmap (后續(xù)也實現(xiàn)這兩個的一步出圖)等畫圖方法,具體看這個教程http://guangchuangyu.github.io/2016/01/go-analysis-using-clusterprofiler/或Biobabble公眾號逐哈。
這個意見得重視,不過大夏天的问顷,鍋還是不能背著昂秃,回應下Y叔的回應。
一個command出圖杜窄,小白上點心可以的
圖是ggplot2畫的(散點圖)肠骆,在腳本功能描述里有寫。只需要在終端輸入腳本的名字sp_enrichmentPlot.sh
(腳本需在環(huán)境變量中)就可以看到腳本功能描述塞耕、輸入文件樣例蚀腿、參數(shù)列表和使用例子。
目前腳本的輸入是不支持Windows的Excel格式的扫外,只支持TAB分割的文本文件莉钙。之前一直在Linux下工作,文件名都不寫后綴的筛谚。后來跟Windows用戶打交道多磁玉,為了方便他們打開,強行加了xls后綴驾讲,這一點有誤導蚊伞。
腳本依賴R席赂,這是應該做一個判斷和提示的。但有R卻提示ggplot2包或其它包不存在厚柳,是可以用-i TRUE來自動安裝包的氧枣。(最近在跟德春合作,完善包的自動檢測和安裝别垮,最后一起整合在分發(fā)包便监。)
Y叔的這兩點確實戳中了當前文檔信息不完善的弱點,也希望大家多反映使用過程中的問題碳想,幫助我們改進文檔烧董。
不過話說回來,如果仔細看了當前腳本的功能描述和參數(shù)提示胧奔,上點心得小白不只可以一步畫圖逊移,還可以隨意調(diào)整樣式。
不只clusterprofiler的用戶需要
如果用clusterprofiler做富集分析龙填,write.table輸出結果胳泉,那么輸入文件、R岩遗、ggplot2都有了扇商,一步出圖沒有問題。(Windows下沒bash宿礁?https://mp.weixin.qq.com/s/g1twNEsPWZb_tZFDyoTqVQ案铺,看這里)
Y叔說既然有了clusterprofiler,一步步在R里面做不是很好梆靖,為啥要把數(shù)據(jù)導出來控汉?
首先,轉換數(shù)據(jù)返吻、存儲數(shù)據(jù)這一步是獨立于作圖的姑子。圖只能顯示部分數(shù)據(jù)(這點Y叔也有提到,用了simplify會好些测僵,但也還會有不少通路)街佑,所有富集條目導出作為文章附表,以顯示信息的全面和真實恨课。
其次,即便可以畫出所有富集數(shù)據(jù)(用一副大圖)岳服,也會先對結果做下篩選剂公,一些特別基礎的、極父層的生物富集通路也會選擇不展示吊宋,優(yōu)先展示樣品屬性更相關的纲辽。畢竟是看在哪些通路里面富集,不是看在哪些通路里面最富集。所以需要導出數(shù)據(jù)拖吼,做下篩選鳞上,然后一步繪圖。
再次吊档,不少人拿到的是已經(jīng)做好的富集分析結果(可能是clusterprofiler的篙议,也可能是其它軟件的),總不能再導入clusterprofiler繪圖吧怠硼,可以有更簡單的方式的鬼贱。
最后,一個圖做出來香璃,需要反復修改这难。今天用clusterprofiler做了富集分析,運用dotplot配合不同參數(shù)出了圖葡秒;過幾天心情一變姻乓,想換個風格了,怎么辦眯牧?再運行一次clusterprofiler還是加載之前存儲的.Rdata蹋岩。好像都不太方便,還是用導出的文本一步出圖吧炸站。
關于硬傷
示例圖中沒有overlap不是繪圖腳本的問題星澳,是數(shù)據(jù)篩選的問題。腳本如實反映篩選出的數(shù)據(jù)旱易。如果選擇的通路在一個基因集合富集禁偎,另外一個不富集,就不顯示阀坏;如果選擇的通路是在多個基因集中都富集的如暖,但富集程度不同,是可以顯示的;而且這些通路會優(yōu)先顯示在圖的上部忌堂,下面我會給出例子盒至。
一步出圖也可以定制
說到互動,一步出圖不只可以士修,而且還可以記錄互動(給每個輸出文件加個唯一的字符串做為文件名的一部分就可以了枷遂,不過這個沒用到,之前就沒寫這個參數(shù))棋嘲。
拿Y叔的數(shù)據(jù)做個例子
library(clusterProfiler)
library(dplyr)
data(gcSample)
x <- compareCluster(gcSample,'enrichDO')
#獲取數(shù)據(jù)酒唉,取出每個組最富集的10個條目,存儲起來
#不麻煩的吧
x <- x@compareClusterResult
y = x %>% group_by(Cluster) %>%top_n(-10, pvalue)
y = x[x$Description %in% y$Description,]
在樣品名中包含差異基因的數(shù)目
#這個不解釋了沸移,前面繪圖教程中提到過
y$Sample <- paste0(y$Cluster,"(",unlist(lapply(strsplit(y$GeneRatio, '/'), function(x)x[2])),")")
#這次存成tsv痪伦,省得誤會了
write.table(y,file="enrichment.tsv", sep="\t", col.names=T, row.names=F,quote=F)
首先出第一個圖侄榴,很簡單,一個命令网沾,設置下顏色癞蚕。
+更正下,我這里作弊了一下辉哥,之前點的大小都是Gene Count這樣的純數(shù)字列來顯示的桦山,不支持GeneRatio這樣的分數(shù)形式的列(上一版本,如果GeneRatio出現(xiàn)在橫軸是支持的)证薇,做了下修改度苔,也支持了。如果想用之前的代碼重復浑度,把-s GeneRatio改為-s Count寇窑。
#具體參數(shù)解釋看上一篇公眾號文章
# -C:指定顏色,默認是紅綠箩张,可以是任何兩個R支持的顏色名字
sp_enrichmentPlot.sh -f enrichment.tsv -oSample -T string -v Description -c p.adjust -s GeneRatio -C"'red','blue'" -w 16
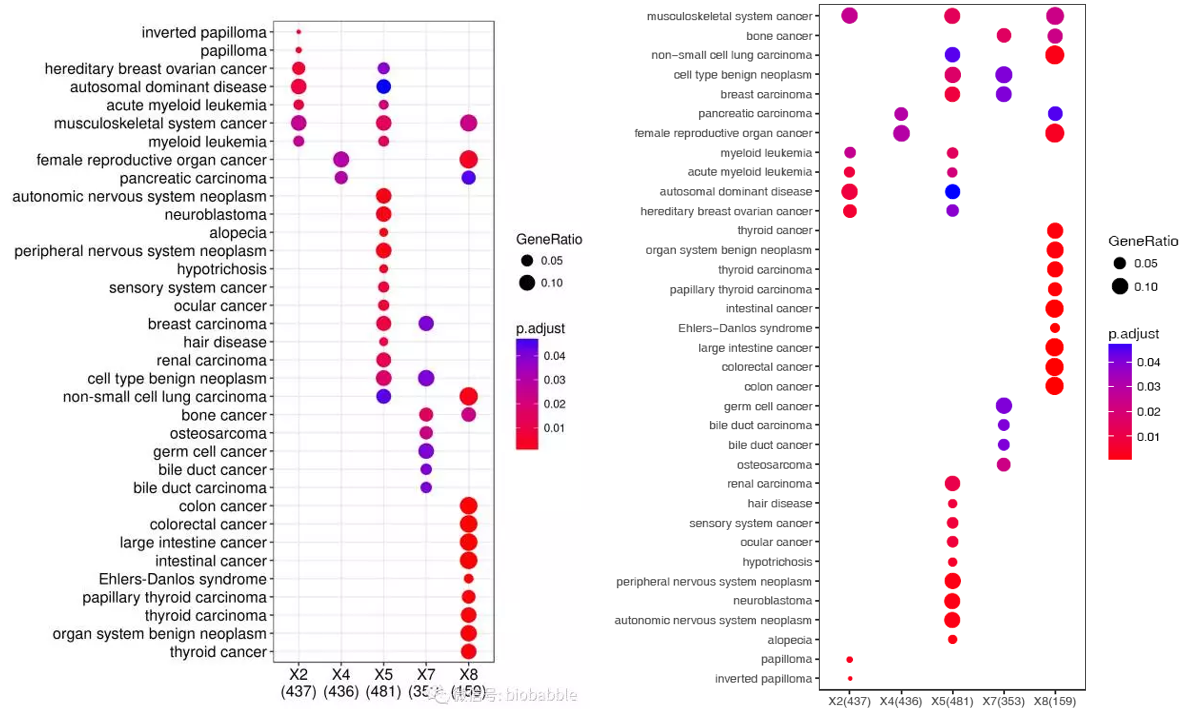
圖略有不同,是因為排序的方式不同先慷。sp_enrichmentPlot.sh設置的是先按照出現(xiàn)在不同樣品最多的條目優(yōu)先的策略饮笛,可以清晰的看到哪些是不同基因集共同富集的,哪些是不同基因集特異富集的论熙。其它的都一樣福青。當然沒有設置字體參數(shù),字體略小了些脓诡。
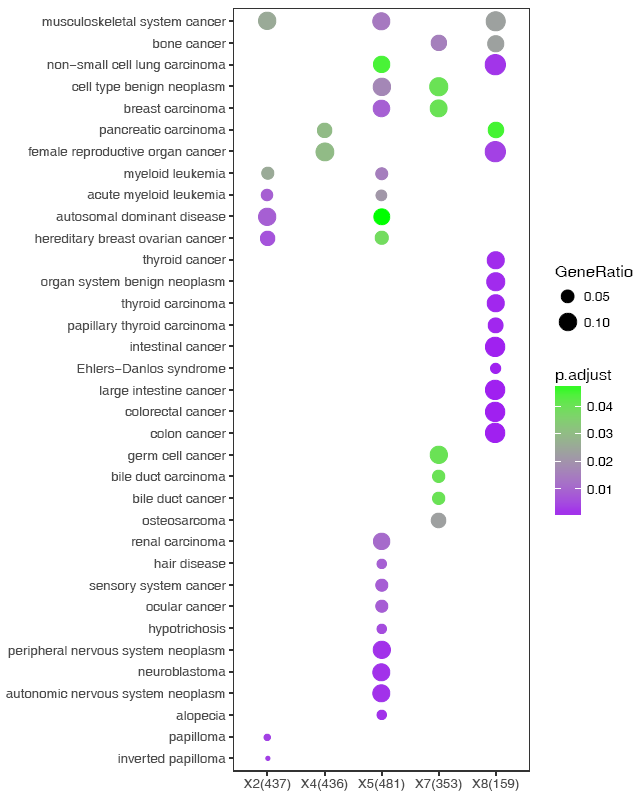
說還想要個紫綠的无午,也可以,只需改兩個單詞祝谚,而不是加一段代碼scale_color_continuous(low='purple',
high='green')宪迟。
sp_enrichmentPlot.sh -f enrichment.tsv -oSample -T string -v Description -c p.adjust -s GeneRatio -C"'purple','green'" -w 16
改變下形狀,也可以啊交惯。程序中都留有一個參數(shù)可以寫入ggplot2語句次泽,就是下面的-z。那么席爽,ggplot2能修改的樣式也都可以意荤。常用的修改會做成一個參數(shù),不常用的就只能直接寫命令了只锻。能在Rstudio里面敲玖像,那么也能在命令行敲。
#-z:后跟任何合法的ggplot2語句
sp_enrichmentPlot.sh -f enrichment.tsv -oSample -T string -v Description -c p.adjust -s GeneRatio -C"'purple','green'" -w 16 -z "+aes(shape=GeneRatio>0.1)"
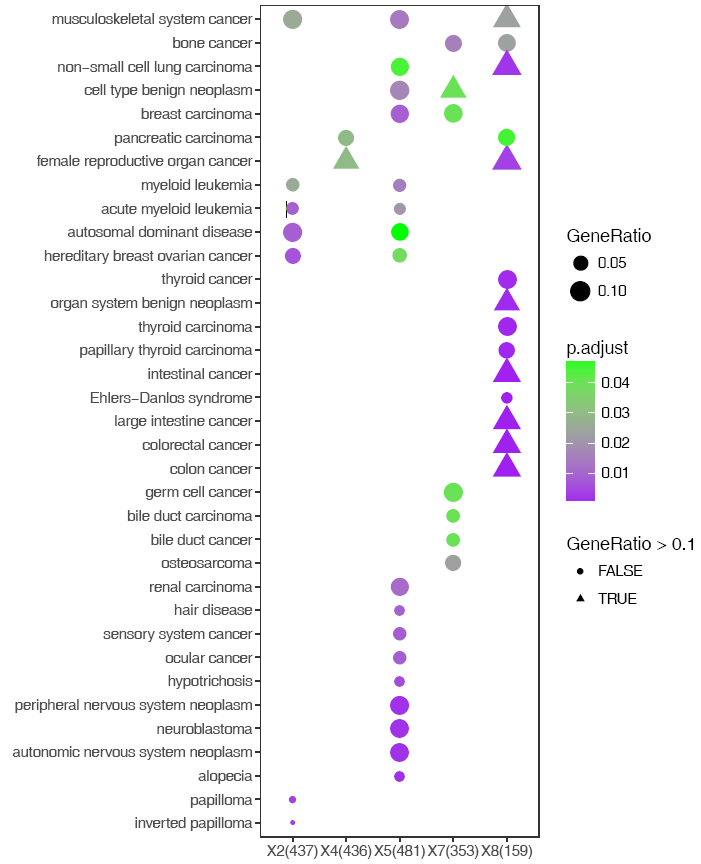
-z “+ any legal ggplot2command”
當然這一步也可以不通過增加ggplot2語句來實現(xiàn)炬藤,在數(shù)據(jù)中加一列就好了御铃,通過-S指定參數(shù)。
不加ggplot2語句,只修改命令行參數(shù)沈矿,sp_enrichmentPlot.sh也可以調(diào)整圖例的位置上真、輸出文件的格式、是否對p.adj取log羹膳、是否分面等睡互。
一步作圖的優(yōu)勢
一步作圖相比于直接寫R代碼有什么好處呢?
1.模塊化好陵像。也就是Y叔提到的數(shù)據(jù)處理和可視化分開就珠;一步作圖,只是作圖醒颖,不做無關的處理妻怎,更隨意。
2.易用性強泞歉。敲代碼逼侦,總不如給改數(shù)來得快。
3.重用性強腰耙。假如我要做十幾個分開的基因集合呢榛丢?一段段復制代碼?改錯了或忘記改某個地方了怎么辦挺庞?
4.快速出圖晰赞。先快速出個原型,再接著調(diào)整选侨。