tags: Trimmomatic NGS fastq
NGS 原始數(shù)據(jù)過濾對(duì)后續(xù)分析至關(guān)重要,去除一些無用的序列也可以提高后續(xù)分析的準(zhǔn)確率和效率彤灶。Trimmomatic 是一個(gè)功能強(qiáng)大的數(shù)據(jù)過濾軟件瞧壮。
Trimmomatic 介紹
Trimmomatic 發(fā)表的文章至今已被引用了 2810 次,是一個(gè)廣受歡迎的 Illumina 平臺(tái)數(shù)據(jù)過濾工具。其他平臺(tái)的數(shù)據(jù)例如 Iron torrent 痊硕,PGM 測(cè)序數(shù)據(jù)可以用 fastx_toolkit 、NGSQC toolkit 來過濾押框。
Trimmomatic 支持多線程岔绸,處理數(shù)據(jù)速度快,主要用來去除 Illumina 平臺(tái)的 Fastq 序列中的接頭橡伞,并根據(jù)堿基質(zhì)量值對(duì) Fastq 進(jìn)行修剪盒揉。軟件有兩種過濾模式,分別對(duì)應(yīng) SE 和 PE 測(cè)序數(shù)據(jù)兑徘,同時(shí)支持 gzip 和 bzip2 壓縮文件刚盈。
另外也支持 phred-33 和 phred-64 格式互相轉(zhuǎn)化,現(xiàn)在之所以會(huì)出現(xiàn) phred-33 和 phred-64 格式的困惑挂脑,都是 Illumina 公司的鍋(damn you, Illumina!)藕漱,不過現(xiàn)在絕大部分 Illumina 平臺(tái)的產(chǎn)出數(shù)據(jù)也都轉(zhuǎn)為使用 phred-33 格式了。
Trimmomatic 過濾步驟
Trimmomatic 過濾數(shù)據(jù)的步驟與命令行中過濾參數(shù)的順序有關(guān)崭闲,通常的過濾步驟如下:
- ILLUMINACLIP: 過濾 reads 中的 Illumina 測(cè)序接頭和引物序列肋联,并決定是否去除反向互補(bǔ)的 R1/R2 中的 R2。
- SLIDINGWINDOW: 從 reads 的 5' 端開始刁俭,進(jìn)行滑窗質(zhì)量過濾橄仍,切掉堿基質(zhì)量平均值低于閾值的滑窗。
- MAXINFO: 一個(gè)自動(dòng)調(diào)整的過濾選項(xiàng)牍戚,在保證 reads 長度的情況下盡量降低測(cè)序錯(cuò)誤率侮繁,最大化 reads 的使用價(jià)值。
- LEADING: 從 reads 的開頭切除質(zhì)量值低于閾值的堿基翘魄。
- TRAILING: 從 reads 的末尾開始切除質(zhì)量值低于閾值的堿基鼎天。
- CROP: 從 reads 的末尾切掉部分堿基使得 reads 達(dá)到指定長度。
- HEADCROP: 從 reads 的開頭切掉指定數(shù)量的堿基暑竟。
- MINLEN: 如果經(jīng)過剪切后 reads 的長度低于閾值則丟棄這條 reads斋射。
- AVGQUAL: 如果 reads 的平均堿基質(zhì)量值低于閾值則丟棄這條 reads育勺。
- TOPHRED33: 將 reads 的堿基質(zhì)量值體系轉(zhuǎn)為 phred-33叨橱。
- TOPHRED64: 將 reads 的堿基質(zhì)量值體系轉(zhuǎn)為 phred-64烁登。
Trimmomatic 簡單用法
由于 Trimmomatic 過濾數(shù)據(jù)的步驟與命令行中過濾參數(shù)的順序有關(guān)褂乍,因此料按,如果需要去接頭狐蜕,建議第一步就去接頭尾抑,否則接頭序列被其他的過濾參數(shù)剪切掉部分之后就更難匹配更難去除干凈了芭商。
單末端測(cè)序模式
在 SE 模式下语盈,只有一個(gè)輸入文件和一個(gè)過濾之后的輸出文件:
java -jar <path to trimmomatic jar> SE [-threads <threads>] [-phred33 | -phred64] [-trimlog
<logFile>] <input> <output> <step 1> <step 2> ...
-trimlog 參數(shù)指定了過濾日志文件名哑了,日志中包含以下四列內(nèi)容:
- read ID
- 過濾之后剩余序列長度
- 過濾之后的序列起始?jí)A基位置(序列開頭處被切掉了多少個(gè)堿基)
- 過濾之后的序列末端堿基位置
- 序列末端處被剪切掉的堿基數(shù)
由于生成的 trimlog 文件中包含了每一條 reads 的處理記錄赘方,因此文件體積巨大(GB 級(jí)別),如果后面不會(huì)用到 trim 日志弱左,建議不要使用這個(gè)參數(shù)窄陡。
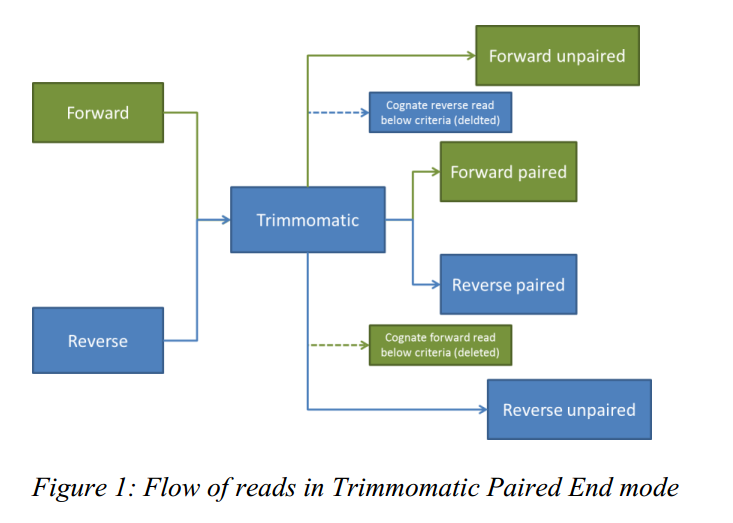
雙末端測(cè)序模式
在 PE 模式下,有兩個(gè)輸入文件拆火,正向測(cè)序序列和反向測(cè)序序列跳夭,但是過濾之后輸出文件有四個(gè),過濾之后雙端序列都保留的就是 paired们镜,反之如果其中一端序列過濾之后被丟棄了另一端序列保留下來了就是 unpaired 币叹。
java -jar <path to trimmomatic.jar> PE [-threads <threads] [-phred33 | -phred64] [-trimlog
<logFile>] >] [-basein <inputBase> | <input 1> <input 2>] [-baseout <outputBase> |
<paired output 1> <unpaired output 1> <paired output 2> <unpaired output 2> <step 1> <step 2> ...
其中 -phred33 和 -phred64 參數(shù)指定 fastq 的質(zhì)量值編碼格式,如果不設(shè)置這個(gè)參數(shù)模狭,軟件會(huì)自動(dòng)判斷輸入文件是哪種格式(v0.32 之后的版本都支持)颈抚,雖然軟件默認(rèn)的參數(shù)是 phred64,如果不確定序列是哪種質(zhì)量編碼格式胞皱,可以不設(shè)置這個(gè)參數(shù)邪意。
輸入輸出文件
PE 模式的兩個(gè)輸入文件:sample_R1.fastq sample_R2.fastq以及四個(gè)輸出文件:sample_paired_R1.clean.fastq sample_unpaired_R1.clean.fastq sample_paired_R1.clean.fastq sample_unpaired_R1.clean.fastq
通常 PE 測(cè)序的兩個(gè)文件,R1 和 R2 的文件名是類似的反砌,因此可以使用 -basein 參數(shù)指定其中 R1 文件名即可雾鬼,軟件會(huì)推測(cè)出 R2 的文件名,但是這個(gè)功能實(shí)測(cè)并不好用宴树,因?yàn)檐浖荒茏詣?dòng)識(shí)別推測(cè)三種種格式的 -basein:
- Sample_Name_R1_001.fq.gz -> Sample_Name_R2_001.fq.gz
- Sample_Name.f.fastq -> Sample_Name.r.fastq
- Sample_Name.1.sequence.txt -> Sample_Name.2.sequence.txt
建議不用 -basein 參數(shù)策菜,直接指定兩個(gè)文件名(R1 和 R2)作為輸入。
輸出文件有四個(gè)酒贬,當(dāng)然也可以像上文一樣指定四個(gè)文件名又憨,但是參數(shù)太長有點(diǎn)麻煩,有個(gè)省心的方法锭吨,使用 -baseout 參數(shù)指定輸出文件的 basename蠢莺,軟件會(huì)自動(dòng)為四個(gè)輸出文件命名。例如 -baseout mySampleFiltered.fq.gz 零如,文件名中添加 .gz 后綴躏将,軟件會(huì)自動(dòng)將輸出結(jié)果進(jìn)行 gzip 壓縮锄弱。輸出的四個(gè)文件分別會(huì)自動(dòng)命名為:
- mySampleFiltered_1P.fq.gz - for paired forward reads
- mySampleFiltered_1U.fq.gz - for unpaired forward reads
- mySampleFiltered_2P.fq.gz - for paired reverse reads
- mySampleFiltered_2U.fq.gz - for unpaired reverse reads
此外,如果直接指定輸入輸出文件名祸憋,文件名后添加 .gz 后綴就是告訴軟件輸入文件是 .gz 壓縮文件会宪,輸出文件需要用 gzip 壓縮。
過濾原理
每一步的過濾如果需要多個(gè)參數(shù)蚯窥,通常用冒號(hào):將各個(gè)參數(shù)隔開掸鹅,當(dāng)然參數(shù)的先后順序是有要求的。
ILUMINACLIP
從名字可以看出拦赠,這一步是為了去除 illumina 接頭的巍沙,這個(gè)軟件其實(shí)就是專為 illumina 平臺(tái)數(shù)據(jù)而設(shè)計(jì)的。
為了更好理解測(cè)序 reads 中為什么會(huì)有引物和接頭序列矛紫,我畫了一個(gè)文庫加上接頭之后的結(jié)構(gòu)示意圖赎瞎,也把引物結(jié)合部位大概標(biāo)了出來:
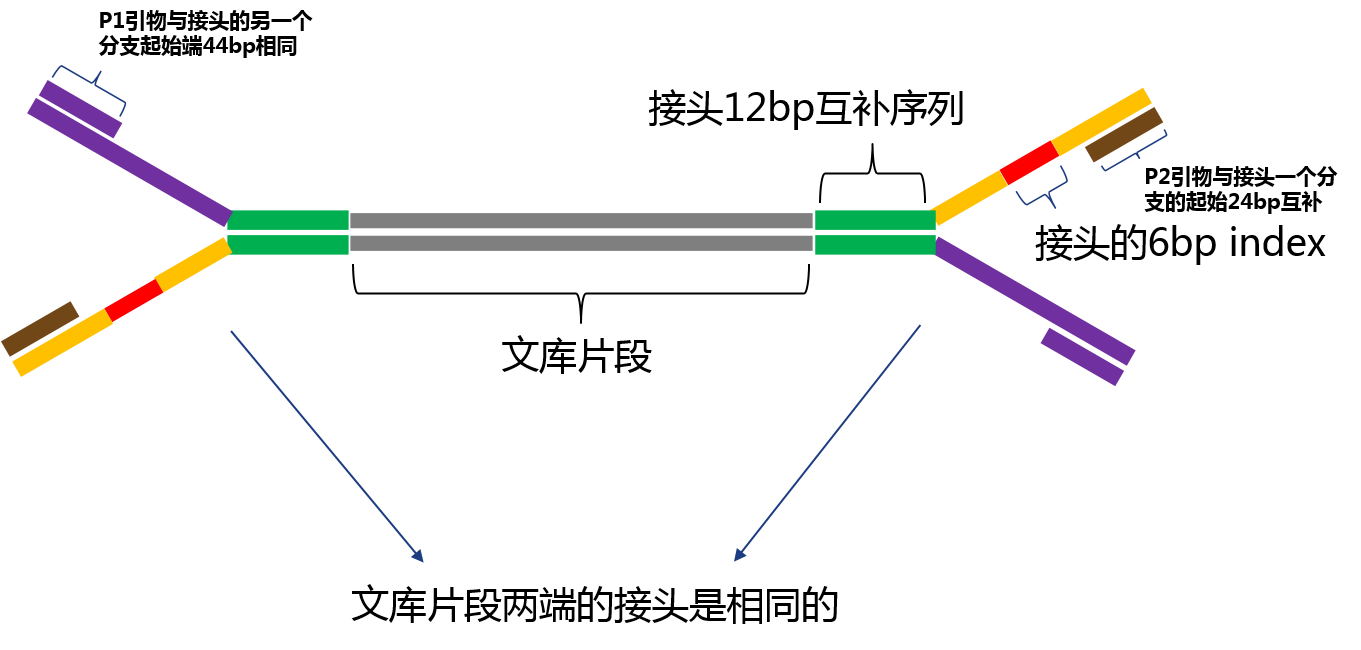
這個(gè)文庫結(jié)構(gòu)示意圖理解之后就容易理解測(cè)序過程了。
去除接頭以及引物序列看似簡單颊咬,但需要權(quán)衡靈敏度(保證接頭和引物去除干凈)和特異性(保證不是接頭和引物的序列不被誤切除),由于測(cè)序中可能存在的隨機(jī)錯(cuò)誤讓去接頭這樣一個(gè)簡單的操作變的復(fù)雜牡辽。
雖然理論上接頭序列和引物序列可能出現(xiàn)在 reads 中的任何位置喳篇,但實(shí)際上序列中出現(xiàn)接頭和引物大部分情況下都是由于文庫插入片段比測(cè)序讀長短導(dǎo)致的,這種情況在 reads 的開頭部分是有一段可用序列的态辛,末端包含了接頭的全長或部分序列麸澜,如果末端只有接頭的一部分序列,那么去除這殘缺的接頭序列也不是容易的事奏黑。
然而炊邦,在 PE 測(cè)序模式下如果文庫的插入片段比測(cè)序讀長短,那么 read1 和 read2 中非接頭序列的那部分會(huì)完全反向互補(bǔ)熟史,Trimmomatic 有一個(gè) ‘palindrome’ 模式會(huì)利用這個(gè)特點(diǎn)進(jìn)行接頭序列的去除馁害。
下圖中 A、B蹂匹、C碘菜、D 四種情況就是 Trimmomatic 去除接頭和引物的四種模式:
- 紅色條形:被切除的序列
- 綠色條形:保留下來的有效讀長
- 深藍(lán)色條形:接頭序列
- 淺藍(lán)色條形:引物序列
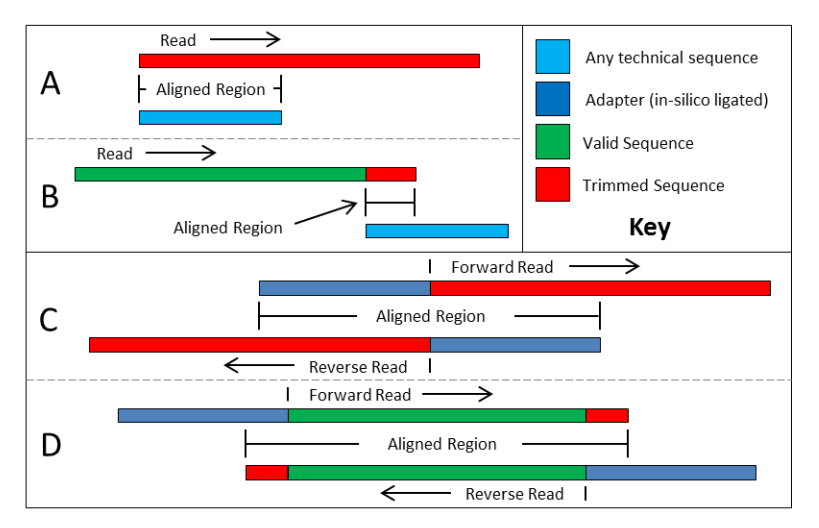
A 模式:測(cè)序 reads 從起始位置開始就包含了完整的接頭序列,那么根據(jù) Illumina 測(cè)序原理限寞,這整條 reads 都不可能包含有用序列了忍啸,整條 reads 被丟棄。
B 模式:這種相對(duì)常見履植,由于文庫插入片段比測(cè)序讀長短计雌,會(huì)在 reads 末端包含部分接頭序列,若是這部分接頭序列足夠長是可以識(shí)別并去除的玫霎,但如果接頭序列太短凿滤,比接頭匹配參數(shù)設(shè)置的最短長度還短传泊,那么就無法去除。但是鸭巴,如果是 PE 測(cè)序眷细,可以按照 D 模式去除 reads 末端的很短的接頭序列。
C 模式:PE 測(cè)序可能出現(xiàn)這種情況鹃祖,正向測(cè)序和反向測(cè)序有部分完全反向互補(bǔ)溪椎,但是空載的文庫,兩個(gè)接頭直接互連恬口,這樣的 reads 不包含任何有用序列校读,正反向測(cè)序 reads 都被丟棄。
D 模式:是 Trimmomatic 利用 PE 測(cè)序進(jìn)行短接頭序列去除的典范祖能,如果文庫插入片段比測(cè)序讀長短歉秫,利用正反向測(cè)序 reads 中一段堿基可以完全反向互補(bǔ)的特點(diǎn),將兩個(gè)接頭序列與 reads 進(jìn)行比對(duì)养铸,同時(shí)兩條 reads 之間也互相比對(duì)雁芙,可以將 3' 末端哪怕只有 1bp 的接頭序列都可以被準(zhǔn)確去除,相對(duì) B 模式去除接頭污染更徹底钞螟。
Trimmomatic 使用了一種類似序列比對(duì)軟件(例如 Isaac aligner兔甘,一個(gè)超快速的 alignment 軟件)的兩步策略來搜索潛在的接頭序列。首先鳞滨,使用接頭序列中的一段種子序列(seed 長度不超過 16bp)與測(cè)序 reads 進(jìn)行比對(duì)洞焙,如果種子序列在測(cè)序 reads 中有足夠好的比對(duì)結(jié)果(具體由 seedMismatch 參數(shù)決定),就啟動(dòng)第二步的接頭全長與 reads 比對(duì)拯啦。第一步的 seed 搜索速度很快澡匪,可以過濾掉沒有接頭污染的 reads ,這種兩步搜索的方法使得接頭序列的查找效率很高褒链。
在第二步的接頭序列和測(cè)序 reads 全長比對(duì)統(tǒng)計(jì)比對(duì)分值時(shí)唁情,罰分策略考慮了測(cè)序堿基的質(zhì)量值Q,每一個(gè)比對(duì)上的堿基加分 0.6碱蒙,每一個(gè)錯(cuò)配的堿基減分 Q/10荠瘪,考慮堿基質(zhì)量值可以降低低質(zhì)量堿基(高測(cè)序錯(cuò)誤率)錯(cuò)配對(duì)整個(gè)比對(duì)得分的影響。在這個(gè)規(guī)則下赛惩,一段 12bp 的接頭序列完全比對(duì)到 reads 上得分為 7.2, 25bp 的接頭序列完全比對(duì)到 reads 上得分為 15哀墓。因此在 ILLUMINACLIP 參數(shù)中 simple clip threshold 的值建議為 7-15 之間(即上圖中 A/B 比對(duì)模式比對(duì)得分閾值)。
對(duì)于 palindromic 模式的比對(duì)(上圖中 D 模式)喷兼,可以比對(duì)上的序列長度會(huì)更長篮绰,為了保證識(shí)別接頭序列的準(zhǔn)確率,比對(duì)得分的閾值也更高季惯,例如 reads的 R1 和 R2 中有 50bp 序列可以反向互補(bǔ)匹配吠各,得分為 30臀突。這種模式下,Trimmomatic 可以識(shí)別并去除 reads 中非常短的接頭序列贾漏。
ILLUMINACLIP 參數(shù)說明:按照規(guī)定順序候学,ILLUMINACLIP 的參數(shù)列表如下(各個(gè)參數(shù)之間以冒號(hào)分開),PE 測(cè)序需要注意最后一個(gè)參數(shù)纵散。對(duì)于 SE 測(cè)序最后兩個(gè)參數(shù)可以不設(shè)置梳码。
ILLUMINACLIP:<fastaWithAdaptersEtc>:<seed mismatches>:<palindrome clip threshold>:<simple clip threshold>:<minAdapterLength>:<keepBothReads>
ILLUMINACLIP:TruSeq3-SE:2:30:10 #接頭和引物序列在 TruSeq3-SE 中,第一步 seed 搜索允許2個(gè)堿基錯(cuò)配伍掀,palindrome 比對(duì)分值閾值 30掰茶,simple clip 比對(duì)分值閾值 10,palindrome 模式允許切除的最短接頭序列為 8bp(默認(rèn)值)蜜笤,palindrome 模式去除與 R1 完全反向互補(bǔ)的 R2(默認(rèn)去除)
fastaWithAdaptersEtc:指定包含接頭和引物序列(所有被視為污染的序列)的 fasta 文件路徑濒蒋,Trimmomatic 自帶了一個(gè)包含 Illumina 平臺(tái)接頭和引物序列的 fasta 文件,可以直接用這個(gè)把兔。
seedMismatches:指定第一步 seed 搜索時(shí)允許的錯(cuò)配堿基個(gè)數(shù)沪伙,例如 2。
palindrome clip threshold:指定針對(duì) PE 的 palindrome clip 模式下垛贤,需要 R1 和 R2 之間至少多少比對(duì)分值(上圖中 D 模式)焰坪,才會(huì)進(jìn)行接頭切除,例如 30聘惦。
simple clip threshold:指定切除接頭序列的最低比對(duì)分值(上圖 A/B 模式),通常 7-15 之間儒恋。
minAdapterLength:只對(duì) PE 測(cè)序的 palindrome clip 模式有效善绎,指定 palindrome 模式下可以切除的接頭序列最短長度,由于歷史的原因诫尽,默認(rèn)值是 8禀酱,但實(shí)際上 palindrome 模式可以切除短至 1bp 的接頭污染,所以可以設(shè)置為 1 牧嫉。
keepBothReads:只對(duì) PE 測(cè)序的 palindrome clip 模式有效剂跟,這個(gè)參數(shù)很重要,在上圖中 D 模式下酣藻, R1 和 R2 在去除了接頭序列之后剩余的部分是完全反向互補(bǔ)的曹洽,默認(rèn)參數(shù) false,意味著整條去除與 R1 完全反向互補(bǔ)的 R2辽剧,當(dāng)做重復(fù)去除掉送淆,但在有些情況下,例如需要用到 paired reads 的 bowtie2 流程怕轿,就要將這個(gè)參數(shù)改為 true偷崩,否則會(huì)損失一部分 paired reads辟拷。
看一個(gè) PE150 數(shù)據(jù)的測(cè)試,就知道 keepBothReads 參數(shù)的重要性了:
$ java -jar trimmomatic-0.36.jar PE -phred33 F-2-test_R1.fastq.gz F-2-test_R2.fastq.gz -baseout F-2.fastq.gz ILLUMINACLIP:TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:51
ILLUMINACLIP: Using 1 prefix pairs, 0 forward/reverse sequences, 0 forward only sequences, 0 reverse only sequences
Input Read Pairs: 2500 Both Surviving: 1633 (65.32%) Forward Only Surviving: 828 (33.12%) Reverse Only Surviving: 12 (0.48%) Dropped: 27 (1.08%)
TrimmomaticPE: Completed successfully
# 使用 ILLUMINACLIP 默認(rèn)的第六個(gè)參數(shù) false阐斜,只有 65.32% paired reads 保留下來
$ java -jar trimmomatic-0.36.jar PE -phred33 F-2-test_R1.fastq.gz F-2-test_R2.fastq.gz -baseout F-2.fastq.gz ILLUMINACLIP:TruSeq3-PE.fa:2:30:10:8:true LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:51
ILLUMINACLIP: Using 1 prefix pairs, 0 forward/reverse sequences, 0 forward only sequences, 0 reverse only sequences
Input Read Pairs: 2500 Both Surviving: 2439 (97.56%) Forward Only Surviving: 22 (0.88%) Reverse Only Surviving: 16 (0.64%) Dropped: 23 (0.92%)
TrimmomaticPE: Completed successfully
# 將 ILLUMINACLIP 第六個(gè)參數(shù)改為 true衫冻,其余所有參數(shù)均相同,結(jié)果有 97.56% paired reads 保留下來
SLIDINGWINDOW
滑窗剪切谒出,統(tǒng)計(jì)滑窗口中所有堿基的平均質(zhì)量值隅俘,如果低于設(shè)定的閾值,則切掉窗口到推。
SLIDINGWINDOW 參數(shù)如下:
SLIDINGWINDOW:<windowSize>:<requiredQuality>
SLIDINGWINDOW:4:15 #設(shè)置 4bp 窗口考赛,堿基平均質(zhì)量值閾值 15
widowSize:設(shè)置窗口大小
requiredQuality:設(shè)置窗口堿基平均質(zhì)量閾值
MAXINFO
包含一個(gè)可以自動(dòng)調(diào)整的過濾條件,在保留盡可能長的序列和保持序列中堿基測(cè)序錯(cuò)誤率盡可能低之間進(jìn)行平衡莉测,以達(dá)到 trim 之后保留序列的價(jià)值最大化颜骤。
對(duì)于不同的應(yīng)用場(chǎng)景,一條 reads 序列的價(jià)值由以下三個(gè)因素決定:
- 允許的最短 read 長度:只有在測(cè)序 reads 足夠長的情況下捣卤,才能將 reads map 到參考序列的唯一位置上忍抽。過短的序列往往能 map 到參考基因組上的多個(gè)位置(特異性差),這種非特異性的 map 結(jié)果可以給出的有價(jià)值信息很少(對(duì)后續(xù)分析沒什么用)董朝。保證 reads 可以 map 到參考序列的唯一位置上需要的 reads 最短長度也與參考序列本身的長度和序列復(fù)雜度有關(guān)鸠项,但是通常最短 reads 要求不低于 40bp。
- 需求的額外 reads 長度:在保證 reads 可以 map 到參考序列上的唯一位置的情況下子姜, reads 序列越長祟绊,可能對(duì)后續(xù)分析越有利。當(dāng)然哥捕,這也需要看應(yīng)用場(chǎng)景牧抽,對(duì)于 RNA-seq 來說只需要統(tǒng)計(jì)某個(gè)位置 map 上的 reads 數(shù)目,這種情況下 reads 長度只要滿足唯一比對(duì)要求即可遥赚。而對(duì)于 de-novo 序列組裝或者 Resequencing 檢測(cè)變異扬舒,reads 序列長度越長越好,reads 越長可以為后續(xù)分析提供越多的有效證據(jù)凫佛。
- 對(duì)測(cè)序錯(cuò)誤的敏感性:即測(cè)序錯(cuò)誤率對(duì)后續(xù)分析工作是否造成嚴(yán)重影響讲坎,有的應(yīng)用中對(duì) reads 測(cè)序錯(cuò)誤零容忍,就需要非常嚴(yán)格的過濾條件愧薛,而有的應(yīng)用對(duì)測(cè)序錯(cuò)誤不敏感晨炕,就可以使用寬松的過濾條件。
MAXINFO 有兩個(gè)參數(shù)厚满,第一個(gè) target read length 控制上面的因素一府瞄,即允許的最短 read 長度。第二個(gè)參數(shù) strictness 是控制因素二和因素三之間的平衡,即滿足最短 read 長度的情況下遵馆,是保留盡可能多的堿基鲸郊,還是保證盡可能低的測(cè)序錯(cuò)誤率。
MAXINFO 過濾從 reads 3' 端開始進(jìn)行剪切货邓,在考慮上述三個(gè)因素的情況下統(tǒng)計(jì)所有可能的 trim 方式的到的 clean reads 的 INFO 分值(即所謂的 reads 價(jià)值)秆撮,這三個(gè)因素分別以不同的方式影響最終的 reads INFO 分值:
- 最短 read 長度:最終保留的 reads 長度與 INFO 分值之間是 logistic 函數(shù)關(guān)系,即 INFO 分值與 reads 長度之間是 S 形增長曲線(類似大腸桿菌生長曲線)换况,大概相當(dāng)于职辨,當(dāng)保留下來的 reads 長度小于最短長度時(shí),INFO 分值隨著 reads 長度增加呈指數(shù)級(jí)增長戈二,當(dāng)保留下來的 reads 長度達(dá)到最短 read 長度要求之后舒裤,INFO 分值的增長會(huì)變緩。
- 超出最短 read 長度的部分:INFO 分值會(huì)隨著額外的 reads 長度增加了線性增長觉吭,線性系數(shù)與 MAXINFO 的第二個(gè)參數(shù)有關(guān)腾供,為 1 - strictness。
- 測(cè)序錯(cuò)誤敏感性:最終保留下來的所有堿基的質(zhì)量值都被用來計(jì)算 reads 包含測(cè)序錯(cuò)誤的概率鲜滩,出現(xiàn)測(cè)序錯(cuò)誤的概率提升會(huì)降低 INFO 分值伴鳖,錯(cuò)誤概率對(duì) INFO 分值的影響權(quán)重與 strictness 相關(guān)。
針對(duì)一條 read 的任何可能的剪切方式都計(jì)算出 INFO 分值徙硅,最終的 reads 長度和切除的堿基由 INFO 最大值決定榜聂。實(shí)際上這三個(gè)影響因子作用的方式不同:
- 當(dāng) reads 長度比最短 read 長度還短時(shí) INFO 分值被序列長度主導(dǎo),因?yàn)樘痰?reads 根本無法提供足夠的有用信息嗓蘑。
- 當(dāng) reads 長度滿足最短 read 長度要求時(shí)须肆,reads 的長度因素就不再是影響 INFO 分值的主導(dǎo)因素了,而且一旦 reads 長度足夠長時(shí)桩皿,由于 logistic 函數(shù)的關(guān)系休吠,reads 長度就不怎么貢獻(xiàn) INFO 分值了。
- 超出最短 read 長度要求的堿基對(duì) INFO 貢獻(xiàn)有限业簿,而另一方面,由于堿基的增加導(dǎo)致 reads 中出現(xiàn)測(cè)序錯(cuò)誤的概率增大阳懂,這會(huì)導(dǎo)致對(duì) INFO 的罰分梅尤。控制這超出最短 read 長度的序列對(duì) INFO 分值的影響到底是罰分還是得分岩调,就看 strictness 參數(shù)了巷燥。
- 對(duì)于大部分足夠長的 reads,保留超出最低要求的堿基序列是利還是弊号枕,會(huì)依照堿基質(zhì)量值分布和 strictness 參數(shù)來決定如何 trim缰揪。
參數(shù)說明:
MAXINFO:<targetLength>:<strictness>
targetLength:使得 reads 可以 map 到參考序列上唯一位置的最短長度(likely)。
strictness:一個(gè)介于 0 - 1 之間的小數(shù),決定如何平衡 最大化 reads 長度 或者 最小化 reads 出現(xiàn)錯(cuò)誤的概率钝腺,當(dāng)參數(shù)設(shè)置小于 0.2 時(shí)傾向于最大化 reads 長度抛姑,當(dāng)參數(shù)設(shè)置大于 0.8 時(shí)傾向于最小化 reads 中出現(xiàn)測(cè)序錯(cuò)誤的概率。
LEADING
從 reads 的起始端開始切除質(zhì)量值低于設(shè)定的閾值的堿基艳狐,直到有一個(gè)堿基其質(zhì)量值達(dá)到閾值定硝。
LEADING:<quality>
quality:設(shè)定堿基質(zhì)量值閾值,低于這個(gè)閾值將被切除毫目。
TRAILING
從 reads 的末端開始切除質(zhì)量值低于設(shè)定閾值的堿基蔬啡,直到有一個(gè)堿基質(zhì)量值達(dá)到閾值。Illumina 平臺(tái)有些低質(zhì)量的堿基質(zhì)量值被標(biāo)記為 2 镀虐,所以設(shè)置為 3 可以過濾掉這部分低質(zhì)量堿基箱蟆。官方推薦使用 Sliding Window 或 MaxInfo 來代替 LEADING 和 TAILING。
TRAILING:<quality>
quality:設(shè)定堿基質(zhì)量值閾值刮便,低于這個(gè)閾值將被切除空猜。
CROP
不管堿基質(zhì)量,從 reads 的起始開始保留設(shè)定長度的堿基诺核,其余全部切除抄肖。一刀切,把所有 reads 切成相同的長度窖杀。
CROP:<length>
length:reads 從末端除之后保留下來的序列長度
HEADCROP
不管堿基質(zhì)量漓摩,從 reads 的起始開始直接切除部分堿基。
HEADCROP:<length>
length:從 reads 的起始開始切除的堿基數(shù)
MINLEN
設(shè)定一個(gè)最短 read 長度入客,當(dāng) reads 經(jīng)過前面的過濾之后管毙,如果保留下來的長度低于這個(gè)閾值,整條 reads 被丟棄桌硫。被丟棄的 reads 數(shù)會(huì)被統(tǒng)計(jì)在 Trimmomatic 日志的 dropped reads 中夭咬。
MINLEN:<length>
length:可被保留的最短 read 長度
TOPHRED33
此選項(xiàng)可以將過濾之后的 Fastq 文件中質(zhì)量值這一行轉(zhuǎn)為 phred-33 格式。
TOPHRED64
此選項(xiàng)可以將過濾之后的 Fastq 文件中質(zhì)量值這一行轉(zhuǎn)為 phred-64 格式铆隘。
接頭序列和引物序列
Trimmomatic 也可以自己制作包含接頭和引物序列的 fasta 文件卓舵,格式可以參考軟件自帶的 adapters 文件夾中的格式。
adapters 文件夾中包含 illumina 測(cè)序 TruSeq2膀钠,TruSeq3 針對(duì) SE 和 PE 的通用接頭和引物序列掏湾。