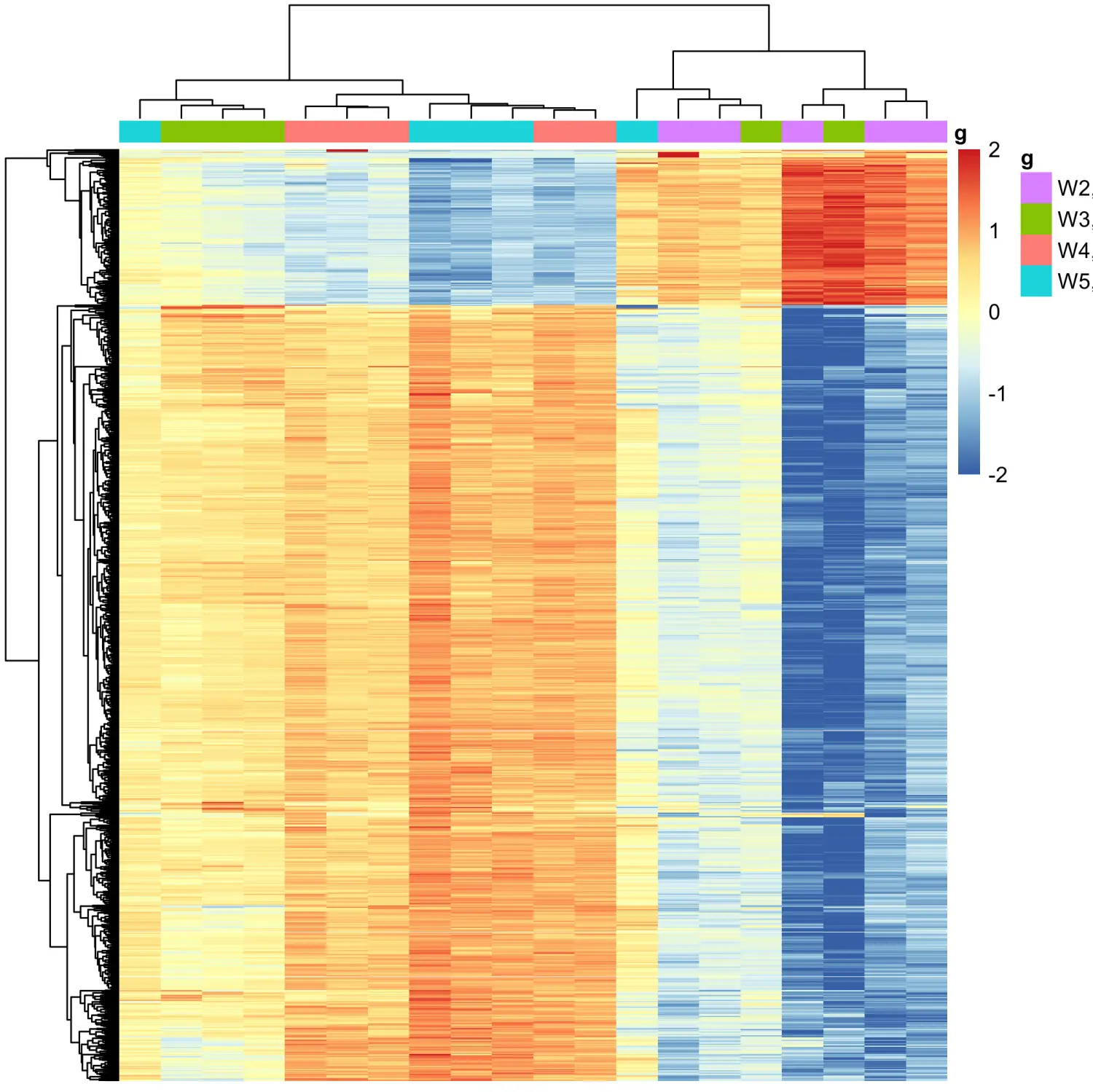
本次配置的環(huán)境是Linux版本的R studio server明也。直接install_github("jmzeng1314/AnnoProbe")安裝出現(xiàn)installatio...
-
解決Linux Rstudio server 安裝AnnoProbe報(bào)錯(cuò)
-
-
獨(dú)立開(kāi)發(fā)者,終身學(xué)習(xí)者,喜歡交朋友顽频,歡迎討論,共同成長(zhǎng)太闺,一起進(jìn)步糯景。
-
@ACh_20d2 根據(jù)提供的教程,應(yīng)該是cor_dist
#' Generates a distance matrix using the square root of (1 - spearman).
#'
#' @param dat.mat Matrix of data (features X samples).
#' @return Distance structure.
#' @export
cor_dist <- function(dat.mat) {
dist.mat <- as.dist(sqrt(1 - cor(dat.mat, method = 'spearman')))
return(dist.mat)
}基于單細(xì)胞測(cè)序的蛋白活性推斷(PISCES)分析流程筆記,凎蟀淮!
寫(xiě)在前面 感謝califano-lab[https://github.com/califano-lab]團(tuán)隊(duì)將代碼無(wú)私的分享出來(lái)=最住。=! 本流程需要一點(diǎn)點(diǎn)R語(yǔ)言與linux基...
-
請(qǐng)問(wèn)各位大佬怠惶,“RegProcess('pisces-1-net-final.tsv', mat1, out.dir = 'tutorial/', out.name = 'pisces-1-net-')” 這里的mat1是不是 pisces1 <- readRDS('pisces-clust_aracne-mats/pisces-1-arac.rds')涨缚?。如果是策治,在運(yùn)行過(guò)程中連續(xù)出現(xiàn)WARNING! NOT CONVERGENT! number of iterations= 1000 是哪里出了問(wèn)題脓魏?
基于單細(xì)胞測(cè)序的蛋白活性推斷(PISCES)分析流程筆記,凎通惫!
寫(xiě)在前面 感謝califano-lab[https://github.com/califano-lab]團(tuán)隊(duì)將代碼無(wú)私的分享出來(lái)=轧拄。=! 本流程需要一點(diǎn)點(diǎn)R語(yǔ)言與linux基...
-
不看私信<br>看不到的文章是被簡(jiǎn)書(shū)鎖了
-
周運(yùn)來(lái)俐末,男,長(zhǎng)大了才會(huì)遇到的帥哥奄侠,穩(wěn)健卓箫,瀟灑,大方垄潮,靠譜烹卒。一段生信緣,一棵技能樹(shù)弯洗。生信技能樹(shù)核心成員旅急,單細(xì)胞天地供稿人,簡(jiǎn)書(shū)創(chuàng)作者牡整,單細(xì)胞數(shù)據(jù)科學(xué)家藐吮。
-
公眾號(hào):TOP生物信息<br>B站:TOP菌<br>WeChat:TOPJUN996<br><br>top2讀研谣辞,學(xué)習(xí)方向:癌癥基因組、腫瘤免疫沐扳、單細(xì)胞組學(xué)
-
-
生信阿拉丁纱烘,為實(shí)用而生
-
歡迎大家分享自己在完成生信技能樹(shù)R語(yǔ)言作業(yè)時(shí)的筆記心得 初級(jí)10 個(gè)題目:http://www.bio-info-trainee.com/3793.html 中級(jí)要求是:http://www.bio-info-trainee.com/3750.html 高級(jí)要求是完成20題: http://www.bio-info-trainee.com/3415.html
-
-
-
-
-
收集一些生物信息學(xué)的分析流程的文章渊胸。感謝這些簡(jiǎn)友整理的生信分析流程旬盯! 如果簡(jiǎn)友投稿沒(méi)有被我收入專題,可能是我沒(méi)看到或者收入到其他針對(duì)性更強(qiáng)的專題中了翎猛。望見(jiàn)諒胖翰! ???????????????????????? 文章的收錄是有順序的,查看的時(shí)候請(qǐng)點(diǎn)擊“最新收錄”然后進(jìn)行查看切厘。 ?? 全基因組測(cè)序(WGS)分析 ?? 轉(zhuǎn)錄組分析RNA-seq的分析流程 ?? 染色質(zhì)免疫共沉淀技術(shù)ATAC-Seq萨咳、ChIP-Seq分析 ?? 單細(xì)胞scRNA-seq分析 ?? 癌癥的預(yù)后標(biāo)志物查找 ?? 系統(tǒng)發(fā)育演化進(jìn)化樹(shù)構(gòu)建與分析 ?? 群體遺傳學(xué)分析 ?? 全基因組關(guān)聯(lián)分析(GWAS) ?? 基因家族的分析 ?? 甲基化測(cè)序與分析 ?? KEGG/GO功能富集分析 ?? 圖形繪制具體細(xì)節(jié)的比如熱圖、曼哈頓圖的繪制等 ?? python疫稿、perl培他、R的包的使用過(guò)程 ?? 具體工具的使用,比如GATK的使用過(guò)程 ?? 數(shù)據(jù)庫(kù)的挖掘方法遗座,比如TCGA數(shù)據(jù)庫(kù)的使用 ?? 另外就是整個(gè)文章走下來(lái)的流程 ?? ?? ??
-
從0開(kāi)始途蒋,包括軟件安裝腾降,環(huán)境設(shè)置,原始數(shù)據(jù)下載碎绎,轉(zhuǎn)換螃壤,差異基因分析等整個(gè)流程
-
中科院上海生科院碩博連讀生奸晴。<br>
-
生信技能樹(shù)寄啼,生信菜鳥(niǎo)團(tuán)逮光,jimmy